Virus gigantes, carne de pollo sin pollo, la batalla de los CRISPR y un ordenador manejado por un cerebro
Como dijo alguna vez Albert Einstein: “cada día sabemos más y entendemos menos”, y de eso va el post que os traigo hoy: cómo puede ser que algunos virus sean más grandes que bacterias y cómo puede obtenerse carne de pollo… sin necesidad de criar pollos. Pues poder se puede; esto es lo más interesante que he leído esta semana.
Los virus gigantes de las amebas.
Stephen Jay Gould escribió muchísimos trabajos sobre evolución, probablemente muchos no sepáis que era aquel hombrecito de poblado bigote y raya al lado que asesoraba a Lisa Simpson cuando ésta había creído descubrir un ángel. Uno de los microorganismos a los que SJ Gould hace referencia de forma recurrente son las amebas. Éstas son microorganismos unicelulares y eucariotas, es decir tienen un núcleo donde albergan su material genético separado del resto de contenido citoplasmático. Es decir sus células tienen en esencia las mismas características estructurales que las de los humanos, pero se las apañan ellas solas para hacer todas las funciones que nosotros tenemos más o menos dividas y organizadas. Probablemente la ameba más conocida sea Naegleria fowleri o “ameba come-cerebros”, desde luego es la ameba más conocida de Charlie, el profesor de inglés con el que tengo hora y media de conversación a la semana. Tal y como su nombre indica está implicada en casos severos de encefalitis al ser capaz de alcanzar el cerebro humano y, literalmente, merendárselo . SJ Gould sin embargo mencionaba a las amebas como hipotéticos espectadores del proceso evolutivo, en el supuesto de que no hubiera existido la explosión cámbrica, fenómeno que ocurrió hace unos 530 millones de años y en el que, durante tan solo 10 millones de años, se desarrollaron de golpe todos los patrones de diseño de animales que conocemos en la actualidad y que denominamos Filos. Diez millones de años en épocas geológicas es casi un instante, juzgue el lector los 120.000 años que nuestra especie lleva “sólo” en la faz de la tierra. La mayor evidencia paleontológica de esta explosión podemos obtenerla en el yacimiento de Burgess Shale, en la Columbia Británica, y si ésta no hubiera tenido lugar el mundo actual estaría probablemente gobernado por amebas, plantas… y bacterias claro. A continuación unas cuantas fotos de estos seres microscópicos:
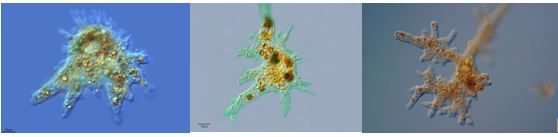
Figura 1: fotografías de amebas al microscopio óptico
Las amebas, sin quererlo, han provocado también un giro en la Virología actual por serendipia, ese término tan de moda que viene a significar “ir a buscar una cosa y encontrar otra mejor de chiripa”. Esto es más o menos así, que me perdone Horace Walpole quien usó el término por primera vez. Resulta que en el año 2003 se descubrieron unos virus específicos llamados mimivirus, que tampoco tendrían mayor importancia de no ser que su mayor peculiaridad es ser gigantes. El descubrimiento de los mimivirus, que infecta a la ameba Acanthamoeba polyphaga (MAAP), fue por tanto accidental y de hecho durante muchos años fue considerado una bacteria intracelular (3). Una de las definiciones de virus puede ser la de “entidades que necesitan otro hospedador para replicarse, carecen de maquinaria metabólica, de producción energética y de replicación, y consisten en una molécula más o menos larga de un ácido nucleico (ADN o ARN de cadena simple o doble) recubierto de una estructura proteica simétrica denominada cápsida”. El descubrimiento de los MAAP originó un debate sobre esta definición, ya que estos virus poseen características tanto de virus como de bacterias. Por ejemplo, los MAAP tienen genomas más largos que algunas bacterias pequeñas, sus cápsulas contienen dos tipos de ácidos nucleicos (ADN y ARN mensajero), además de ciertas moléculas necesarias para la traducción proteica, incluidos los denominados ARN de transferencia (ARNt) y algunas sintetasas de ARNt. Seguramente esto os sonará chino pero el lector avanzado notará en seguida que este fue un descubrimiento sin precedentes dentro no solo de la Virología sino de la Biología.
El aislamiento de los MAAP se llevó a cabo mientras el Dr. Rowbotham realizaba un extenso aislamiento de microorganismos que se caracterizaban por ser intracelulares estrictos en amebas. Si bien la mayor parte de aislados resultaron ser bacterias del género Legionella, fue imposible identificar dos de esos aislados que comenzaron a ser llamados los “cocos de Bradford” (coco, del griego kokkos o baya, hace referencia a una bacteria esferoidal). No fue hasta la secuenciación de su genoma cuando se vio que, sin lugar a dudas, los MAAP eran virus, y de hecho su nombre deriva de la frase MImicking a MIcroorganism (iMItando a un MIcroorganismo). Los MAAP estuvieron “disfrazados” de bacterias a los ojos de los científicos durante un par de décadas.
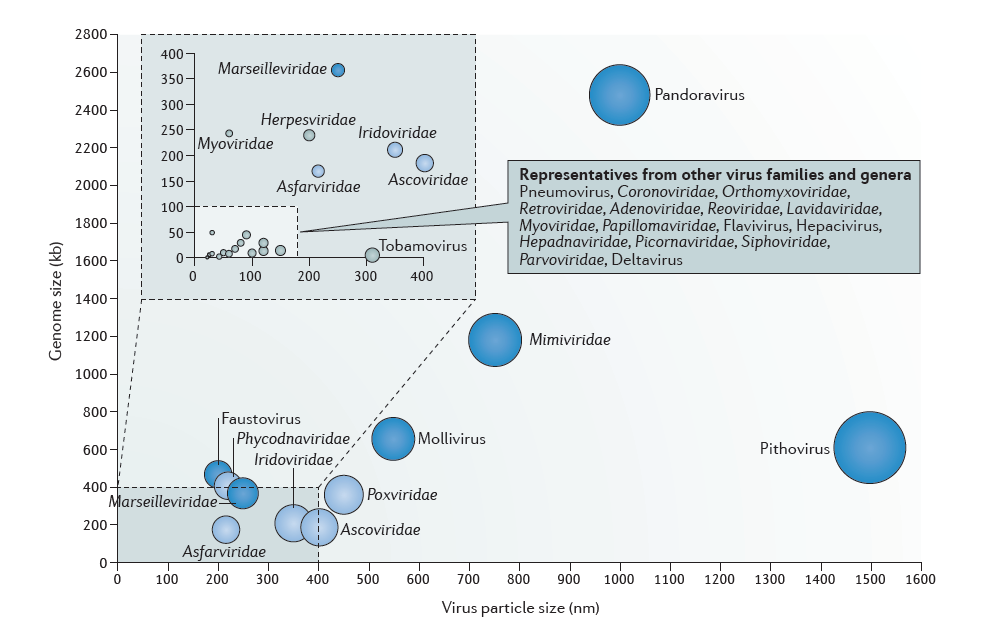
Figura 2. Representación del tamaño de los genomas frente al tamaño de la partícula en distintos virus. Los TRUC, como los MAAP, los Pandoravirus o los Fitovirus, pueden llegar a ser más grande que muchas de las bacterias más conocidas (una bifidobacteria o un lactobacilo tienen un tamaño promedio de una micra o 1000 nm). Figura tomada de Colson, P. y cols. Nature Reviews Microbiology 15, 243-254 (2017).
El origen de los MAAP es bastante incierto, pero se ha propuesto que al tener genes homólogos a bacterias, arqueobacterias y eucariotas estos virus podrían representar la cuarta rama del árbol de la vida, propuesta por Woesse en 1977 y basada en la similitud de las secuencias de ARN ribosomales. Su origen podría entroncarse con el de los primeros eucariotas o protoeucariotas, que serían algo parecido a las actuales amebas. Estos virus gigantes, junto con otros como los Megavirus, son denominados TRUC, del inglés “cosas que resisten una clasificación incompleta” (Things Resisting Uncompleted Classification). La existencia de esta cuarta rama del árbol de la vida es sujeto de controversia, al no existir metodología para cuantificar cuál podría haber sido la transferencia horizontal de genes desde otros microorganismos a los MAAP, y por tanto sigue sujeto a debate.
Carne de pollo… ¿¿¿sin pollos???
Desde hace unos años es posible disponer de carne artificial, igual de gustosa (eso dicen sus creadores) que su contrapartida animal, pero al alcance de pocos bolsillos. Y todo ello sin tener que criar, sacrificar o despiezar un solo animal. Este tipo de carne es el producto estrella de la start-up americana Memphis Meats, quienes ya contaban en su carta con albóndigas de carne, y que acaban de presentar dos nuevos productos: carne de pollo y carne de pato.
¿Cómo se consigue esta carne artificial? Pues en biorreactores celulares específicos como los que hace unos meses nos describía Alberto Morán en su artículo sobre fotocopiadoras celulares. Digamos que el procedimiento o la receta podría ser esta:
- coja usted sus células madre pluripotenciales o células que pueden dar lugar a otros tipos celulares. Nótese la diferencia con una célula madre totipotencial, la cual puede dar lugar a cualquier tipo de célula. No se confunda al comprar este ingrediente.
- disponga un caldo de cultivo con los nutrientes y vitaminas adecuados en las proporciones correctas. No se pase con los oligoelementos que pueden ser tóxicos.
- sazone con las señales moleculares para que las células crezcan y se diferencien de una forma adecuada.
- Cocine a fuego muy muy lento (37ºC) en una atmósfera con un 5% de CO2 y tras 4 a 6 semanas de cocción voilà, su pechuga de pollo (o de pavo) estará lista.
El objetivo de Memphis Meats es ofrecer a sus clientes carne de forma sostenible y sin tener que recurrir a la ganadería extensiva ni al sacrificio de animales, y creen que pueden tenerlo listo para 2021. Por delante les quedan cuatro años para reducir costes, y pasar de los 9000 € aproximadamente que cuesta producir medio kilo de carne de pollo a los 10 € / kilo con los que pretenden entrar (y revolucionar) el comercio de carne a través del canal de distribución de los supermercados. Todo un reto tecnológico por delante.
La batalla de los CRISPR
Hace un par de años Alberto Morán también nos describía en este blog lo que son los CRISPR . No voy a entrar en muchos detalles de lo que son, pero por repasar rápidamente el concepto, se trata de una especie de sistema de defensa procariota que ha tenido la peculiaridad de haber sido adoptado para editar genomas eucariotas. En concreto Alberto nos hablaba del sistema CRISPR-Cas9, capaz de sustituir una secuencia de ADN con una mutación por otra con la secuencia correcta; una especie de copia-pega molecular.
Ahora que los lectores saben de lo que hablo, lo que probablemente recordarán era la batalla legal que por aquel entonces se había desatado en torno a la patente de esta tecnología entre el Broad Institute en Cambridge (BIC) y la Universidad de California en Berkeley (UCB). Esta disputa comenzó en 2012, cuando Jennifer Doudna (UCB) presentó una patente en la que proponía el uso del sistema CRISPR-Cas9 para la edición de genomas bacterianos. Meses más tarde, Feng Zhang (BIC) presenta una patente en la que se protege el uso del sistema CRISPR-Cas9 para la edición de genomas eucariotas, mucho más complejos. A partir de ese momento, la UCB ha tratado de frenar y echar por tierra esta patente alegando que sus investigadores habían desarrollado la tecnología primero, a pesar de que como decimos la aplicación iba dirigida a bacterias y no a eucariotas. De hecho la investigación básica que había dado lugar a este descubrimiento se había descubierto 25 años antes, en España, pero eso es cuestión de un post futuro. Pues bien, hace un mes la Oficina Americana de Patentes y Marcas (USPTO) ha realizado una decisión histórica al defender la serie de patentes que habían sido concedidas originalmente al BIC. Cuando todo parecía estar del lado de la UCB… vuelta a empezar.
La tecnología CRISPR-Cas9 tiene potenciales aplicaciones en biotecnología, medicina o agricultura, entre la que se encuentran opciones tan controvertidas desde el plano ético como es la modificación genética y dirigida de embriones humanos. En este sentido las Academias Naciones de Ciencias y Medicinas de los EEUU ha autorizado recientemente esta técnica si con ella pueden prevenirse enfermedades o discapacidades graves. Puede imaginarse así un futuro en el que no sólo hayamos eliminado de nuestra sociedad muchas enfermedades infecciosas, sino algunas de las mutaciones genéticas responsables de algunas de las enfermedades hereditarias más devastadoras. Y cómo aquí hay negocio, ni el BIC ni la UCB dejarán pasar la oportunidad de generar miles de millones de divisas en beneficios a futuros. (Starling, S. Nature Reviews Microbiology doi:10.1038/nrmicro.2017.21)
Ordenadores manejados por cerebros
La última noticia de ciencia (por esta semana) gira en torno a la última locura de Elon Musk. Tras TESLA, SpaceX e HyperLoop, este extravagante y visionario emprendedor nos presenta Neuralink, start-up que surge con la misión de desarrollar interfaces para poder manejar ordenadores con nuestro cerebro a través de conexiones directas entre ambos a través de “lazos neuronales” y la implantación de electrodos; algo así ocurre en la película Trascendence, protagonizada por Johnny Depp. Además de aplicaciones evidentes, como sería el hecho de que yo pudiese estar escribiendo estas líneas simplemente con pensarlas, o que una máquina pudiese suplir la discapacidad de una persona (por ejemplo unos brazos), detrás de esta iniciativa se esconden desarrollos mucho más complejos y ambiciosos. Uno de ellos es que el ser humano pueda alcanzar la inmortalidad descargando sus pensamientos y su personalidad, y quien sabe si su consciencia, como si de una copia de seguridad se tratase. Incluso alguno podría pensar en una humanidad futura que es inmortal descargando todo el contenido del cerebro y su función cognitiva a modo de sistema operativo.
Y esto ha sido todo en esta semana, aunque los científicos leemos muchas cosas de nuestra especialidad, siempre se encuentra hueco para informar sobre otras cosas muy interesantes. Y como todo buen trabajo, ahí os dejo las referencias en las que me he basado.
About the Author: Borja Sánchez
One Comment
Leave A Comment Cancelar la respuesta
Este sitio usa Akismet para reducir el spam. Aprende cómo se procesan los datos de tus comentarios.








[…] …….https://www.dciencia.es/virus-gigantes-carne-de-pollo-sin-pollo-la-batalla-de-los-crispr-y-un-ordena…https://naukas.com/2012/12/27/el-misterio-de-los-virus-gigantes/https://www.agenciasinc.es/Entrevistas/El-descubrimiento-de-los-virus-gigantes-fue-revolucionario […]